 [Imágenes: Freepik]
[Imágenes: Freepik]
Cuando entramos a LinkedIn, es habitual que nos recomiende conectar con personas que conocemos, ya sea porque hemos estado en la misma universidad o hemos coincidido en la misma empresa. Sin embargo, siempre hay alguna sugerencia que nos sorprende. Por ejemplo, cuando el algoritmo nos recomienda a nuestro cuñado, aunque trabajemos en ámbitos totalmente distintos. Entonces, ¿cómo es posible que LinkedIn sepa que “en la vida real” sí nos conocemos?
Los algoritmos de inteligencia artificial que impulsan estas recomendaciones utilizan una tecnología específica conocida como Graph Neural Networks o redes neuronales gráficas, que se basa en grafos, estructuras formadas por nodos y aristas que los conectan. En el contexto de una red social como la de LinkedIn, se puede generar un grafo donde los nodos representan a cada usuario, mientras que las aristas corresponden a las conexiones entre ellas.
Estos algoritmos recopilan información del entorno inmediato de cada nodo, es decir, de nuestras conexiones directas en LinkedIn. Después agrupan esa información y la integran en el nodo original.
Así, tras este proceso, cada perfil cuenta con una representación actualizada que refleja tanto sus propios datos como los de su red más cercana. Este proceso puede llevarse a cabo varias veces. Así, en la segunda iteración, cuando agreguemos información de nuestros vecinos, estos a su vez ya tendrán agregada información de sus propios vecinos y, en consecuencia, tendremos información del segundo vecindario.
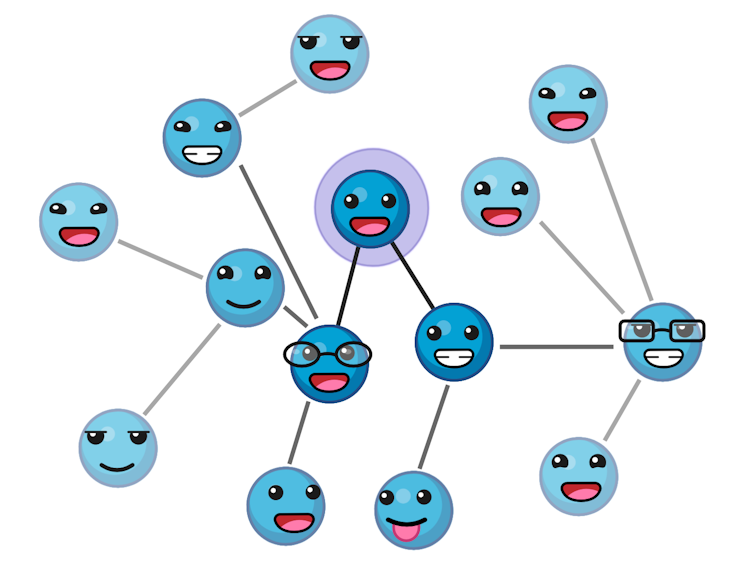
Un entramado de relaciones
Por ello, en estas redes no solo importa toda nuestra información personal, sino con quién hayamos conectado y con quién han conectado nuestras conexiones. En la versión completa del algoritmo de LinkedIn, tal y como se usa en la práctica, no solo existen nodos que representan a los miembros de la red social, también existen otros tipos de nodos, como empresas o publicaciones.
De esta forma, el algoritmo puede obtener información tanto de nuestras conexiones personales como de los contenidos que hemos marcado como favoritos o con los que hemos interactuado.
Por eso, si alguien tiene a su hermana como conexión y además ha dado “me gusta” a publicaciones que le gustan igualmente a su cuñado, el algoritmo detecta que no solo comparten intereses similares, sino que además están conectados personalmente de alguna manera.
¿Y si pudiésemos usar este tipo de algoritmos en biomedicina?
A día de hoy, desarrollar un medicamento desde cero es un proceso muy costoso, tanto en el tiempo que requiere como en la inversión de capital. Muchas veces, se visualiza el proceso de descubrimiento como un embudo donde, por la parte más ancha, entran todos los posibles candidatos y, tras diferentes estadios de investigación, queda solo uno para entrar en ensayo clínico, que (con suerte) lo pasará y estará disponible para su uso médico en la población.
Esto ha llevado a que, en las últimas décadas, haya ganado fama el reposicionamiento de medicamentos: no se busca diseñar uno nuevo, sino encontrar nuevos usos para los ya existentes.
Dianas de proteínas
Generalmente, para tratar una enfermedad, nos enfocamos en atacar a las proteínas responsables. Existen bases de datos públicas y bien documentadas que contienen información sobre qué proteínas ataca cada medicamento. En los últimos años, estas bases de datos han crecido considerablemente.
Como ejemplo, una de las más empleadas, DrugBank, pasó de 841 fármacos aprobados en su primera versión (2006), a 2,751 en su última actualización (2024). Esta reciente disponibilidad de datos permite usar modelos más complejos.
Así, como comentábamos anteriormente, podemos crear una red donde los nodos son medicamentos y proteínas, y las aristas son las interacciones entre ellos registradas en bases de datos. Una vez que tenemos la red, podemos aplicar un algoritmo donde todos los medicamentos se les agregue la información (bioquímica) de las proteínas con las que tuvieron interacción a través de conexiones conocidas.
Con esta información, el modelo puede decirnos las probabilidades de que exista una interacción fármaco-proteína que a priori no teníamos registrada en la base de datos. Los algoritmos pueden analizar de forma eficiente grandes volúmenes de información. Posteriormente, estas interacciones se validan en laboratorio, lo que permite ahorrar tiempo y dinero.
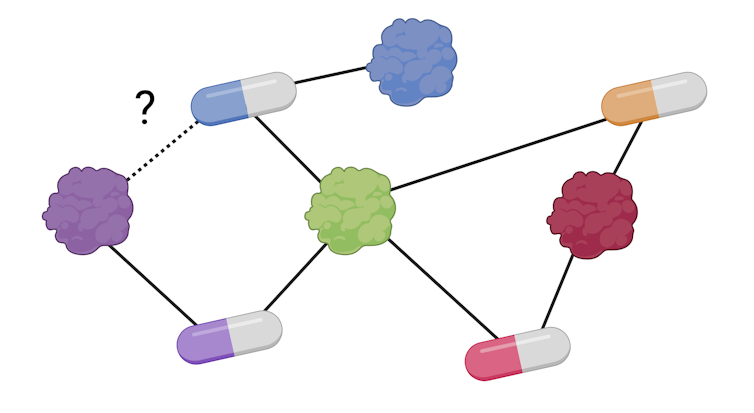
Nuestro aporte
En nuestro laboratorio de Biología Computacional y Genómica Traslacional de la Universidad de Navarra, hemos seguido esta idea para desarrollar GeNNius, un modelo cuyo algoritmo que considera una red entre medicamentos y proteínas. Con su implementación, hemos mejorado los modelos existentes, especialmente, en el tiempo de ejecución: en tan solo un minuto podemos evaluar alrededor de 23,000 interacciones.
Sin embargo, aunque el modelo presenta buenas capacidades de predicción, todavía hay margen de mejora. Por ejemplo, uno de los desafíos surge al evaluar posibles interacciones con moléculas que no forman parte de la red o sobre las que se conoce muy poco. Aunque técnicamente es posible hacerlo, el modelo suele ofrecer resultados con baja confianza en estos casos.
Salvando dichos obstáculos y con más investigación, estos modelos podrían evolucionar en el futuro hacia sistemas que ofrezcan recomendaciones personalizadas para cada paciente.
Mikel Hernaez es director del programa de Biología Computacional y Genómica Traslacional en la Universidad de Navarra. Uxía Veleiro es investigador predoctoral en la misma universidad.
Este artículo fue publicado en The Conversation. Lee el original.

![[Imagen: cortesía de Penguin Random House]](https://fc-bucket-100.s3.amazonaws.com/wp-content/uploads/2026/03/09115101/p-1-91501856-penguin-random-house-branding.webp)
![[Foto: Apple]](https://fc-bucket-100.s3.amazonaws.com/wp-content/uploads/2026/03/09115735/p-1-91502546-macbook-neo-apple-affordable-tech-brand-laptop-services-revenue.webp)
![[Imagen: Freepik]](https://fc-bucket-100.s3.amazonaws.com/wp-content/uploads/2026/03/09123722/Radiografia-Emprendimiento-mujeres-Fast-Company-Mexico-Freepik.jpg)




![[Imagen impulsada por IA]](https://fc-bucket-100.s3.amazonaws.com/wp-content/uploads/2026/03/09084414/geopolitica-moverse-aplastarse.jpg)